Missing Value
อะไรหายไปไหน
Missing Value คือค่าที่สูญหายไปในชุดข้อมูลของเราค่ะ ซึ่งค่าสูญหายนี้มีสาเหตุต่างกัน เช่น สูญหายเพราะ responder ไม่อยากตอบ หรือหายตัวไปกลางครัน (เช่น ถ้าทำแบบสอบถามออนไลน์แล้ว internet หลุดไป) หรือเป็นเพราะผู้วิจัยไม่ได้ให้ตอบคำถามในบางกลุ่ม
Missing value เหล่านี้ทำให้การประมาณค่าของพารามิเตอร์ (see : Distribution in population) ผิดไป และยังส่งผลต่อการยอมรับหรือปฏิเสธสมมติฐานว่าง (null hypothesis) ทำให้เกิด Type I error มากขึ้น
ชนิดการการสูญหาย
-
Missing Completely at Random หรือ MCAR จะเป็นการสูยหายแบบที่เกิดจากการสุ่ม (ใช้คำว่าสุ่มจะดูเหมือนผู้วิจัยสุ่ม แต่ไม่ใช่นะคะ random หรือ สุ่มในที่นี้คือไม่ได้เกิดอย่างเป็นระบบ ไม่มี pattern ในตัวแปรนั้น)
-
Missing at Random (MAR) เกิดค่าสูญหายแบบสุ่มเหมือนกัน แต่มาจากตัวแปรอื่นที่มีผลกับตัวแปรที่ต้องการศึกษา
-
Missing not at Random (or Not Missing at Random; MNAR) เกิดได้หลายกรณีรวมถึงการวาดกรอบการทดลองผิดพลาดแล้วไม่ได้เก็บข้อมูลที่มีผลต่อตัวแปรที่ต้องการศึกษามา
ที่สำคัญ คือ เราจะทราบได้อย่างไรคะว่า ข้อมูลสูญหายใดเป็นแบบ MCAR, MAR or, MNAR?
คือไม่ทราบก็ได้ เพราะบางทีนักวิจัยเองก็ไม่ทราบค่ะ
แต่ว่าเราต้องจัดการกับข้อมูลสูญหายให้เหมาะสม เพราะในกรณี 2 ชนิดหลังจะส่งผลทั้งค่าพารามิเตอร์และค่าความคาดเคลื่อนในกลุ่มตัวอย่าง (SE) เลยค่ะ
ที่นี้ แยกกันนิดนึง
ปกติแล้ว สาย Data analytics เมื่อมีชุดข้อมูลเยอะ ๆ เป็น 1,000 - 10,000 ข้อมูล ก็มักจะใช้วิธีการตัดข้อมูลแถวนั้นทิ้งไปเลย ด้วยวิธีการ na.omit()
แต่ถ้าเป็นงานวิจัยที่เราทุ่มแรงเก็บกลุ่มตัวอย่างและข้อมูล 40 - 500 ข้อมูลด้วยความพยายาม จะตัดข้อมูลทิ้งก็อาจจะเสียดายได้ แต่ถ้าเราจะเอาข้อมูลแถวนั้นคงไว้ ก็ต้องเลือกจัดการมันอย่างเหมาะสมค่ะ
การจัดการข้อมูลที่ศูนย์หาย (missing value analysis)
เราจะใช้ข้อมูล coronavirus จาก package coronavirus
library(coronavirus)
data("coronavirus")
head(coronavirus)
## date province country lat long type cases uid iso2 iso3
## 1 2020-01-22 Alberta Canada 53.9333 -116.5765 confirmed 0 12401 CA CAN
## 2 2020-01-23 Alberta Canada 53.9333 -116.5765 confirmed 0 12401 CA CAN
## 3 2020-01-24 Alberta Canada 53.9333 -116.5765 confirmed 0 12401 CA CAN
## 4 2020-01-25 Alberta Canada 53.9333 -116.5765 confirmed 0 12401 CA CAN
## 5 2020-01-26 Alberta Canada 53.9333 -116.5765 confirmed 0 12401 CA CAN
## 6 2020-01-27 Alberta Canada 53.9333 -116.5765 confirmed 0 12401 CA CAN
## code3 combined_key population continent_name continent_code
## 1 124 Alberta, Canada 4413146 North America NA
## 2 124 Alberta, Canada 4413146 North America NA
## 3 124 Alberta, Canada 4413146 North America NA
## 4 124 Alberta, Canada 4413146 North America NA
## 5 124 Alberta, Canada 4413146 North America NA
## 6 124 Alberta, Canada 4413146 North America NA
จากการดูข้อมูลคร่าวๆ ค่าสูญหาย (NA) ดันไปอยู่ในจังหวัด (province) ไม่เป็นไรนะ สมมติว่าเราสร้าง missing value เอง
เราจะใช้การ subset ข้อมูลที่ต้องการเพียง date country และจำนวน case
coronavirus <- coronavirus[, c("date", "country", "cases")]
head(coronavirus[coronavirus$cases == 0,]) #เดี๋ยวเราจะให้จำนวนเคสที่เป็น 0 เป็นข้อมูลสูญหาย
## date country cases
## 1 2020-01-22 Canada 0
## 2 2020-01-23 Canada 0
## 3 2020-01-24 Canada 0
## 4 2020-01-25 Canada 0
## 5 2020-01-26 Canada 0
## 6 2020-01-27 Canada 0
coronavirus$cases[coronavirus$cases == 0] <- NA #ใส่ข้อมูลสูญหายด้วย NA
#which(!complete.cases(coronavirus$case), arr.ind = TRUE) #เรียกดูสักหน่อย
จะดูข้อมูลทั่วโลกก็อาจจะเยอะไป เราจะ subset ให้เหลือแค่ UK และ Thailand เพื่อความสะดวก ทีนี้ เรามาดู pattern ของข้อมูลสูญหายที่เราใส่เข้าไป
Covid_th_UK <- coronavirus[coronavirus$country == "United Kingdom" | coronavirus$country == "Thailand", ] #subset
head(Covid_th_UK)
## date country cases
## 632 2020-01-22 United Kingdom NA
## 633 2020-01-23 United Kingdom NA
## 634 2020-01-24 United Kingdom NA
## 635 2020-01-25 United Kingdom NA
## 636 2020-01-26 United Kingdom NA
## 637 2020-01-27 United Kingdom NA
เมื่อได้ data ที่ทำการ subset ให้เหลือแค่ประเทศ UK และ Thailand แล้ว เราจะใช้คำสั่ง md.pattern()จาก package mice ในการดู pattern ของค่าสูญหาย
library(mice)
##
## Attaching package: 'mice'
## The following object is masked from 'package:stats':
##
## filter
## The following objects are masked from 'package:base':
##
## cbind, rbind
md.pattern(Covid_th_UK)
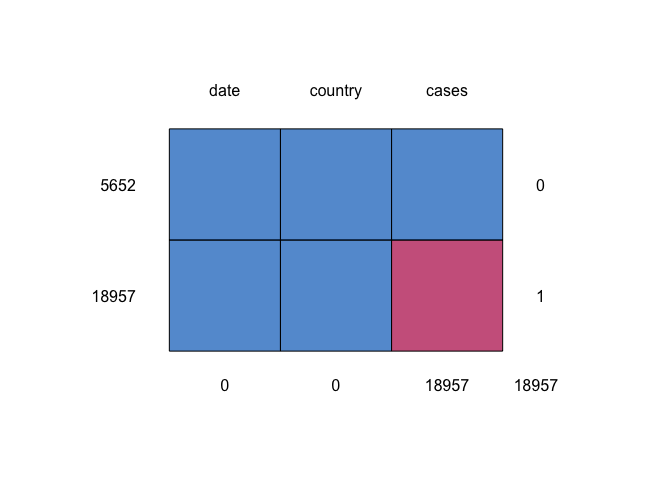
## date country cases
## 5652 1 1 1 0
## 18957 1 1 0 1
## 0 0 18957 18957
เนื่องจากสูญหาย ที่เราใส่ไปมีโอกาสที่จะเป็นแบบ intentionally missed ถ้า dataset ของเรามีขนาดใหญ่พอ เราสามารถที่จะเรียกคำสั่ง na.omit() สำหรับ dataset นั้นได้เลย
Covid_th_UK_omit <- na.omit(Covid_th_UK)
md.pattern(Covid_th_UK_omit)
## /\ /\
## { `---' }
## { O O }
## ==> V <== No need for mice. This data set is completely observed.
## \ \|/ /
## `-----'
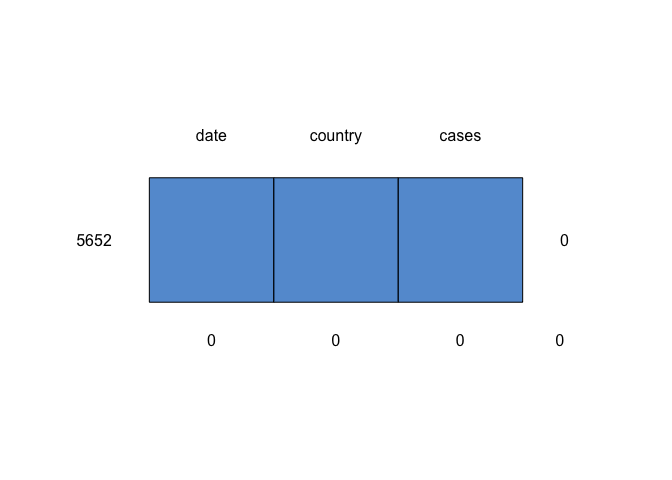
## date country cases
## 5652 1 1 1 0
## 0 0 0 0
แมวบอกเราว่าไม่มี missing value แล้ววววว~
อย่างไรก็ตาม หาก dataset มีขนาดเล็ก โดยเฉพาะกับจากการเก็บข้อมูลแบบทดลองหรือ survey เล็ก ๆ เราอาจจะไม่สามารถตัดค่าสูญหายไปได้ ในกรณีนี้เราสามารถที่จะดูสัดส่วนของข้อมูลที่ศูนย์หายและแทนค่าข้อมูลที่ศูนย์หายนั้น ๆ โดยวิธีการต่างกัน
เช่น
- แทนที่ด้วยค่า Mean : วิธีนี้จะยังทำให้เกิด bias อยู่มากโดยเฉพาะการสูญหายแบบ MAR
- แทนที่ด้วยค่า Mode : วิธีที่ก็ยังจะทำให้เกิด bias ได้กรณที่ scale เป็นมาตรระดับ
- แทนที่ด้วยค่า Median : วิธีนี้ถ้ามีค่าสุดโต่งมาก ๆ ก็ต้องกำจัดค่าสุดโต่งออกก่อน (ด้วยต่างวิธีการมากมาย) แต่ก็ยังทำให้เกิด bias ได้
แล้วทีนี้ หมดที่เรียนมาแล้ว ควรจะทำอย่างไรดี
ทำแบบ 3 วิธีด้านบนได้ค่ะ ถ้าเราเล่าเรียนมาแบบนั้น มันไม่มีผิดนะ แต่ว่ามันจะมี bias ในค่าข้อมูลเกิดขึ้น
แต่เราก็สามารถแทนที่ค่าสูญหายด้วยวิธีอื่นได้ แต่จะมาต่อภาค 2 นะคะ เพราะว่าเริ่มจะเป็นการวิเคราะห์แบบขั้นสูงขึ้นมานิดนึงแล้ว
กลับไปที่ Datastist.com